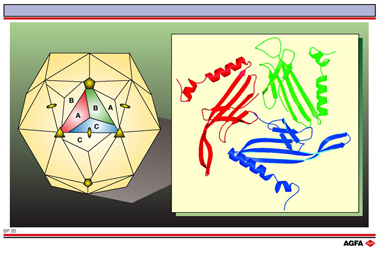
BP25: La structure du bactériophage MS2
|
Obj.: Illustrer une structure biologique complexe: un bactériophage |
Les bactériophages sont des virus qui infectent d’abord des bactéries et qui s’y multiplient par la suite. Un des bactériophages les plus simples, le bactériophage MS2, est un virus à ARN: à l’intérieur de l’enveloppe de la protéine, on trouve un brin d’ARN unique possédant une longueur de 3569 nucléotides. Ce brin d’ARN contient toutes les informations génétiques pour la multiplication du virus.
Etant donné que le génome est ainsi limité, on fait souvent appel à la symétrie pour l’élaboration de l’enveloppe de la protéine. Le bactériophage possède une forme sphérique avec un diamètre d’environ 28 nm, basée sur un icosaèdre. Un icosaèdre est un corps hypothétique constitué par 20 triangles. A gauche, une représentation schématique du bactériophage révèle la structure récurrente – les trois protéines de l’enveloppe A, B et C – de l’enveloppe du virus et un certain nombre d’éléments de symétrie (un pentagone présente un axe quinaire, un triangle présente un axe ternaire et une ellipse présente un axe binaire). A droite, on représente la structure en trois dimensions de ces trois protéines de l’enveloppe (A en rouge, B en vert et C en bleu). Chaque protéine de l’enveloppe est constituée par une série de feuillets b et par deux hélices a. Les trois protéines sont presque identiques. La différence majeure se manifeste dans les boucles entre les structures secondaires. L’ARN est protégé contre le monde extérieur par les feuillets b. Les hélices a jouent un rôle dans le maintien de l’enveloppe de la protéine. Les deux représentations se basent sur des déterminations de la structure cristalline du bactériophage MS2 (référence: Valegard et coll., Nature 345,36(1990), Protein Databank code 2ms2).
Pour l’élaboration des
modèles en trois dimensions basés sur des déterminations de structures
cristallines (illustrations BP7, 9, 13-15, 18, 24-25), on utilise les
programmes MOLSCRIPT (P.J. Kraulis, Appl. Cryst. 24, 946 (1191) et RASTER3D
(E.A. Merritt & M.E.P. Murphy, Acta Cryst. D50, 869 (1994). Des coordinats
sont disponibles via la Protein Databank (F.C. Bernstein et coll., J.
Mol. Biol. 112, 535 (1977),
adresse Internet: http://www.pdb.bnl.gov).